- Inicio
- ⟩
- Tecnología
- ⟩
- Edición de genomas, la técnica que impulsó una revolución científica
Edición de genomas, la técnica que impulsó una revolución científica
La técnica CRISPR es uno de los avances científicos más destacados en la actualidad.
25-03-2019
Por María Luisa Santillán, Ciencia UNAM-DGDC
Una de las aportaciones al conocimiento científico que ha revolucionado y avanzado a pasos agigantados en las últimas décadas, ha sido la técnica conocida como edición de genes.
Su origen parte de la ciencia básica y puede situarse a inicios de la década de los noventa, allá por el año 1993, cuando a partir del estudio del genoma de las bacterias se dieron los primeros descubrimientos de lo que hoy conocemos como tecnología CRISPR.
CRISPR son las siglas de Clustered Regularly Interspaced Short Palindromic Repeats (Repeticiones Palindrómicas Cortas Agrupadas y Regularmente Interespaciadas). Esto significa que hay una parte determinada del genoma de la bacteria en donde hay secuencias repetidas y entre éstas hay unos espaciadores que posteriormente se descubrió que eran secuencias de virus que habían infectado a las bacterias.
Después de años de investigación y de la aportación de distintos grupos en todo el mundo, hoy se tiene una nueva técnica de edición de genes, que consiste en el corte de genes para hacerles modificaciones, ya sea quitándoles un pedazo o colocándoles algún otro elemento.
La doctora Rosa Estela Navarro González, del Instituto de Fisiología Celular de la UNAM, destacó que en la actualidad esta técnica se usa principalmente para investigación básica, por ejemplo, en laboratorio se ha utilizado para demostrar que se puede reparar algún error en un gen que provoca cierta enfermedad.
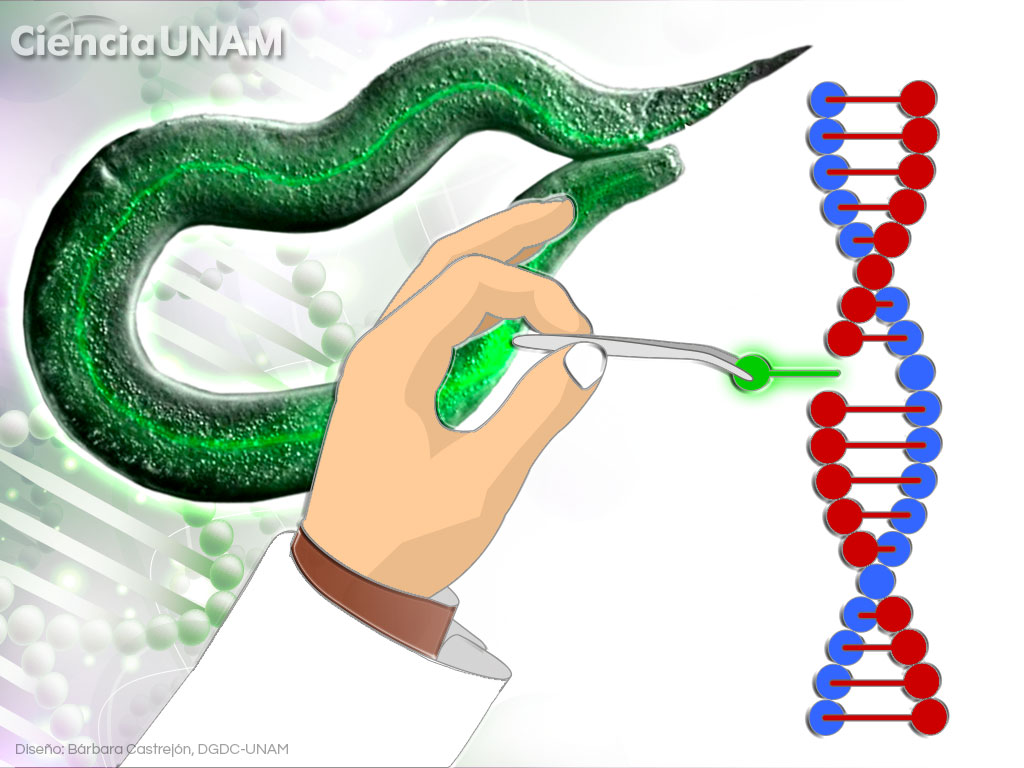
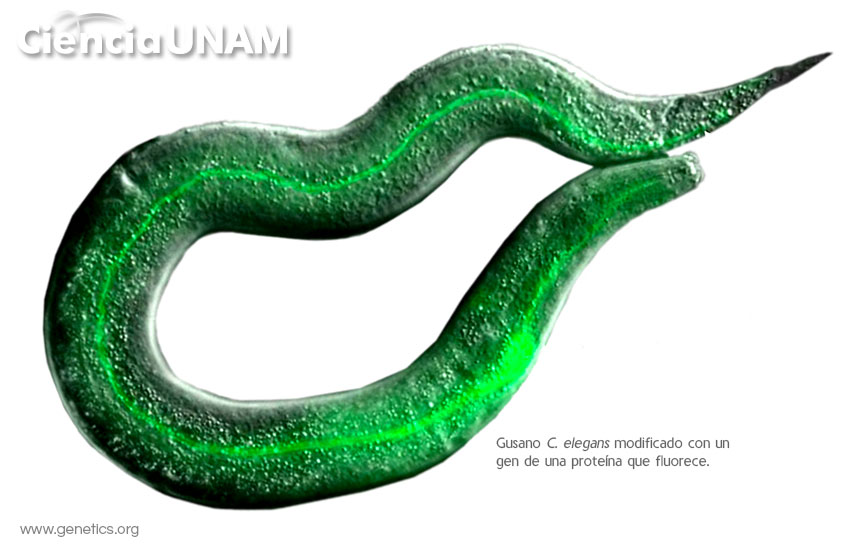
En el laboratorio que ella dirige se estudia un gusano redondo llamado Caenorhabditis elegans. La investigación se enfoca en cómo las células que producen los gametos de ese gusano, es decir, los óvulos y los espermatozoides, se afectan con el estrés.
“Lo que usamos de ese sistema es para poder hacer cortes en el genoma de este gusano y hacer mutaciones. La técnica ha ido evolucionando de tal manera que ahora podemos meter un pedazo de ADN que le sirva para reparar. En nuestro caso, metemos el gen de una proteína que fluorece y el gusano ahora es verde fluorescente y así podemos estudiar en dónde se expresa la proteína”.
Sistema inmune de las bacterias
Para la doctora Navarro, lo interesante de la historia del origen de esta técnica es que surgió haciendo investigación básica, pues el investigador español Francisco Mojica estudiaba bacterias y encontró en su genoma secuencias repetidas y pedazos de virus que infectaban a una bacteria.
Durante la visita de este investigador a México, en una conferencia en El Colegio Nacional, contó que como parte de su proyecto de doctorado pretendía saber cuáles eran los mecanismos por los cuales microorganismos halófitos eran capaces de adaptarse a cambios de salinidad del medio. De ahí partió esta investigación, que dos décadas después sería considerada por la revista Science como el avance científico del 2015.
El doctor Mojica narró cómo empezaron a secuenciar el genoma de estas bacterias, qué tipo de experimentos llevaron a cabo, cómo empezaron a estudiar la expresión de estas secuencias, cuál era su función en estos microorganismos y cómo se leían en distintas condiciones de crecimiento.
Durante una década realizaron distintos experimentos para conocer cuál era la función de estas secuencias. Sin embargo, desde 1987 hasta el año 2004 pasó desapercibido el estudio de estas secuencias repetidas en procariotas, “muy probablemente porque no teníamos ni idea de para qué servían”, dijo.
Fue en 2005 que el Journal Molecular Evolution publicó un artículo en el que se describía cuál era el origen de esas secuencias espaciadoras localizadas entre las repeticiones, pues encontraron que un porcentaje de esos espaciadores coincidían con secuencias de virus que infectaban bacterias.
La doctora Navarro explicó que gracias a la investigación de varios grupos se fue viendo que las bacterias tienen un sistema de defensa en contra de los virus: las infecta un virus, les inyecta su ARN o su ADN y la bacteria toma ese pedazo de genoma y lo mete en su propio genoma. Entonces, ahí hace un corte y mete ese pedazo de gen para acordarse de que la infectó.
De esta manera, la siguiente vez que la infecta, ese virus empieza a producir ARN, el cual se une a una proteína conocida como Cas 9, que es una tijera molecular que lo reconoce, lo busca en el genoma y lo degrada. Es un sistema de memoria que le sirve a la bacteria para defenderse de los virus.
Avances y perspectivas
Esta tecnología ha ido evolucionando poco a poco. Una vez que se conoció para qué servía este mecanismo, grupos de investigación en todo el mundo han aportado conocimientos para el perfeccionamiento de la técnica. Por ejemplo, se dio a conocer la existencia de proteínas Cas (CRISPR-Associated), que son unos genes que se localizan justo al lado de las repeticiones y que hoy se sabe que están asociados a ellas.
Además, se encontró que los espaciadores coincidían con secuencias de virus que infectaban bacterias. También se demostró que las bacterias tienen un sistema de inmunidad adquirida y que las guías de este sistema son lo que se denominó CRISPR-RNA.
Posteriormente, se descubrió la existencia de los PAM (Protospacer Adjacent Motifs), que son secuencias muy cortas que se conservan al lado de los espaciadores; se descubrió que estos motivos PAM eran necesarios para que un sistema CRISPR pudiera actuar sobre un blanco específico, pues lo reconocía guiado por un ARN y la cortaba.
Así como esta tecnología ha servido para realizar distintos avances en materia genómica, también ha sido motivo de distintas controversias.
Tan sólo a principios de diciembre de 2018, el investigador chino He Jiankui dijo haber utilizado la técnica CRISPR para mutar un gen y crear dos bebés genéticamente modificados y resistentes contra el virus del sida.
La controversia no se hizo esperar y la comunidad científica internacional, en su mayoría, reprobó el actuar del investigador. Al respecto la doctora Navarro opina que por ello es urgente legislar en torno a esta tecnología.
Además, la especialista en biología del desarrollo agregó que el riesgo que presenta esta técnica es que todavía le faltan ajustar algunos aspectos, uno de ellos es la especificidad, es decir, que sólo corte donde se le indique.
Entre las principales aplicaciones que se vislumbran de esta técnica en un futuro, se destacan reducir la resistencia de las bacterias a antibióticos, curar enfermedades, además de que se podrá regular la expresión genética.
Además, los pioneros en el desarrollo de esta técnica, como el doctor Mojica, han sido mencionados en la lista de espera para recibir el Premio Nobel. Veremos si en la edición 2019 de los galardones, el fallo finalmente es a favor
Publicaciones relacionadas

Edición del genoma, una revolución irreversible

“Armonía y Revolución”, lema de La Noche de Las Estrellas 2021

En el Día Internacional de la Madre Tierra: Salud de las Plantas, vital para todos